セルソーティング と デジタルPCR のコラボレーション!
このコーナーでは、定期的にバイオ・ラッドの新製品や技術情報のトピックをご紹介します。
多くの生体サンプルは複数の細胞種が混在した状態にあります。 サンプル中のそれぞれの細胞群の遺伝的な背景を理解することで、そのサンプルの、ひいては生体の特徴を理解するのに重要な手がかりが得られます。 例えば、腫瘍中で急速に増殖する細胞亜集団(Subpopulation)を特定し、その特性を知ることは、がんの転移の理解に役立つことでしょう。
今回ご紹介する技術資料では、S3eセルソーターにより、複数の細胞種が混在するサンプルから特定の細胞群を単離し、次いで、QX200ドロップレットデジタルPCRシステムにより、それぞれの細胞群におけるCNV(Copy number variation)解析を行った実験例を示しています。
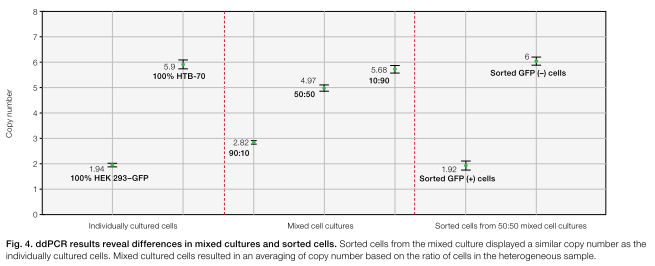
(左)個別に培養したセルラインからのCNV解析。HTB-70は6コピーのサイクリンD1遺伝子(CCND1)を持ち、HEK 293-GFPは2コピー持つ。 (中)先述セルラインを90:10, 50:50, 10:90で混合培養した状態からのCNV解析。それぞれCCND1は見かけ上、2.82コピー、4.97コピー、5.68コピーとして測定される。 (右)50:50の混合培養から99%以上の純度でソーティングした細胞でのCNV解析。それぞれ個別に培養した場合と同等のコピー数として測定された。
本資料では、一方の細胞群としてGFPを発現するセルラインを用いたため、GFPシグナルの有無をマーカーとしてセルソーティングを行いましたが、目的とする実験系によって細胞表面抗原などのマーカーを選択することが可能です。 資料では、S3/S3eセルソーターによるフローサイトメトリー解析の実験データや実験プロトコールも記載されております。詳しくはPDFファイルをご覧ください。
QX200ドロップレットデジタルPCRシステムにおけるCNV解析には、PrimePCR ddPCR Copy Number Assay: Cyclin D1 (CCND1) Human Kitを使用しました。PrimePCR製品の一部につきましては現在国内での取り扱い開始に向けて準備中です。
| Discriminating Copy Number Variation from Heterogeneous Samples Using the S3 Cell Sorter and the QX200 Droplet Digital PCR System |
| S3eセルソーターの製品紹介 |
| バイオラッドの抗体製品 |
QX200 Droplet Digital PCR システムの製品紹介
※当該製品は研究用機器であり、医療機器ではありません。
| 製品紹介:QX200 Droplet Digital PCR システム |
| Droplet Digital PCR 論文データベース |
| Droplet Digital PCR 論文リスト |
| 日本国内のユーザー様からの論文報告のご紹介 |
